Virennachweis mit
geteilter Luciferase
(22.03.2023) Isothermale Amplifikation und CRISPR-Cas sind ein fast perfektes Paar in der Virus-Diagnostik. Noch schlagkräftiger sind sie mit Split-Luciferasen.
Die Gesamtstaatliche COVID-Krisenkoordination (GECKO) in Österreich macht Feierabend. Auch die meisten anderen Länder geben Entwarnung. Aber niemand weiß, ob der Erreger der nächsten Pandemie nicht schon in den Startlöchern steht. Da Infektionskrankheiten aber gerade für schlechter vorbereitete und finanziell weniger gut aufgestellte Länder verheerend sein können, sind einfache und günstige Point-of-care-Diagnostik-Methoden umso wichtiger. Häufig werden für diese isothermale Amplifikationsverfahren wie LAMP (Loop-mediated Isothermal Amplification) und (RT-)Recombinase Polymerase Amplification (RPA) eingesetzt, die unabhängig von teuren qPCR-Geräten funktionieren und eine simple Detektion ermöglichen.
Die Amplifikation bei relativ niedrigen Temperaturen hat aber ihre Tücken. Die bei 65 beziehungsweise 40 Grad Celsius durchgeführten LAMP und RPA sind zu lax, um unspezifische Hybridisierungen völlig zu vermeiden. Ein positives Probenergebnis bedeutet zwar, dass etwas amplifiziert wurde – in vielen Fällen ist es aber nicht die Zielsequenz (Target).
Strategie auf drei Beinen
Die geringen Temperaturanforderungen von LAMP und RPA haben dennoch große Vorteile – vor allem wenn man sie mit Techniken kombiniert, die spezifische von nichtspezifischen Amplifikationen verlässlich unterscheiden können. Hierfür existieren CRISPR/Cas-basierte Verfahren, bei denen Cas12- oder Cas13-Nucleasen gequenchte Fluoreszenz-Sensoren schneiden und dadurch zum Leuchten bringen. Die Methoden sind aber sehr aufwendig und zudem anfällig für Autofluoreszenz.
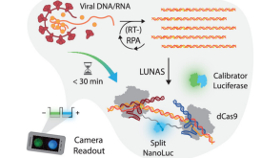
Maarten Merkxens Team von der Eindhoven University of Technology hat sich eine andere Strategie ausgedacht, die auf drei Beinen steht: Sie nutzt RPA als einfache Amplifikationsmethode, setzt auf spezifische Erkennung des Targets durch sgRNAs und ergänzt diese Techniken mit der Komplementierung einer gesplitteten Luciferase. Für Letztere verwendete die Gruppe jedoch nicht das klassische BiFC-System, bei dem ein geteiltes gelbes Fluoreszenzprotein leuchtet, wenn die zwei Komponenten wieder vereint werden. Die niederländischen Forscher und Forscherinnen verwenden stattdessen die zwei Teile der NanoLuc-Luciferase als Sensor.
Stabile NanoLuc-Luciferase
Die Idee für den lumineszenten Nukleinsäure-Sensor (LUNAS) ist nicht ganz neu. Vor fünf Jahren publizierte eine chinesische Gruppe einen dsDNA-Sensor, der auf der dCas9-vermittelten Komplementierung der Split-Firefly-Luciferase basierte (ACS Synth Biol, 6(2):211-6). Die NanoLuc-Luciferase liefert jedoch ein deutlich helleres Signal und ist zudem kleiner und stabiler (ACS Chem Biol, 11(2): 400–8).
Kleines (SB) und großes (LB) Fragment der NanoLuc fusionierte die Gruppe jeweils an den C-Terminus von dCas9 (SB-dCas9 sowie LB-dCas9). Für die Detektion sind zwei spezifische sgRNAs (gRNA A und gRNA B) zuständig. Zusammen mit einem dCas9-Molekül bilden sie zwei unterschiedliche Ribonucleoprotein-Komplexe (RNP). Verbunden durch einen flexiblen Peptidlinker hängen SB und LB von NanoLuc an der dCas9. Binden beide sgRNAs an die Stränge desselben (Ziel-)DNA-Moleküls, kommen sich die Proteinkomponenten genügend nahe, um die NanoLuc zu rekonstituieren. Die komplementierte, enzymatisch aktive NanoLuc wandelt das Substrat Furimazin in ein blau leuchtendes Produkt um.
Alles in einem Pott
Für den Abstand der beiden sgRNA-Bindestellen auf dem Zielmolekül wählte das Team zunächst 12 bis 110 Basenpaare, als optimal erwiesen sich 27 bis 52 Basenpaare. Die PAMs (Protospacer adjacent motifs) befinden sich auf je einem Strang der doppelsträngigen Ziel-DNA und stehen sich gegenüber. Bei dieser Konstellation stehen sich auch die beiden Peptidlinker mit den split-NanoLuc-Komponenten gegenüber und können sich hierdurch genügend nahe kommen.
Zunächst inkubierte das Team die Proben nach der RPA-Reaktion mit dem Sensorgemisch aus RNP und Substrat. Die dazu nötigen 40 Grad Celsius sind für Basenpaarung, Protein- und Substrat-Stabilität aber unproblematisch. Aber warum das Ganze nicht gleich in einem Gefäß im One-pot-Format durchführen? Tatsächlich kamen sich Polymerase, RNP und die anderen Mitspieler nicht in die Quere, denn die Amplifikation ließ sich anhand der zunehmenden Lumineszenz in Echtzeit verfolgen. Die Sache hatte dennoch einen Haken: In der One-pot-Reaktion erhöhte sich zwar anfangs die Intensität der Lumineszenz, sank danach aber wieder. Das ist heikel, wenn die Reaktion nicht von Beginn an verfolgt werden kann, etwa in Point-of-care-Settings. Schuld an der schwächer werdenden Intensität war aber nicht etwa das allmähliche Abdriften der beteiligten Proteine, sondern die allmähliche Depletion des Substrats.
Blaue und grüne Signale
Mit einem Griff in die Trickkiste der Biochemie löste die Gruppe das Problem. Sie setzte dem Test eine zweite NanoLuc zu, die dasselbe Substrat umsetzt, jedoch grünes Licht erzeugt. Anschließend detektierte sie die blauen und grünen Signale. Da das Verhältnis von blau zu grün proportional zur Probenkonzentration ist, konnte das Team auf diese Weise die Konzentration bestimmen.
Das LUNAS-Verfahren der Niederländer hat sich bereits im Labor bewährt. Die Gruppe detektierte mit ihm SARS-CoV-2-Varianten in verschiedenen Proben (Nasen-Rachenraum-Abstrich, Viral-Transport-Medium und Speichel) ohne RNA-Isolation. Die Sensitivität ist mit dem Ct-Wert einer RT-qPCR von etwa 26,5 vergleichbar. Zu konzentriert sollte die Probe nicht sein, denn bei einem massiven dsDNA-Überangebot verteilen sich die sgRNAs großzügig. Einige dsDNAs werden nur von dem SB-Fragment der NanoLuc erreicht, die anderen nur von dem LB-Fragment. Modellsimulationen und Experimente zeigten, dass die Chance für die Komplementierung bei einem Verhältnis von 10 nM dCas9-SB zu 1 nM dCas9-LB am höchsten ist.
Andrea Pitzschke
Van der Veer et al. (2023): Glow-in-the-dark infectious disease diagnostics using CRISPR-Cas9-based split luciferase complementation. ACS Cent Sci, DOI: 10.1021/acscentsci.2c01467
Bild: Van der Veer et al.