Pawlow�sche Colis
(6.2.17) Mit einem lernenden Gen-Schaltkreis E. colis konditionieren? Genau damit traten Zürcher Studenten beim letzten International Genetically Engineered Machine (iGEM)-Wettbewerb in Boston an. Pawlow lässt grüßen – wenigstens theoretisch...
Mit ihrem Projekt erfüllte die Gruppe der ETH Zürich nicht nur die iGEM-Bedingungen für die Goldmedaille, sondern war auch noch nominiert in den Kategorien „Best Part Collection“ und „Best Model“. Und tatsächlich war deren iGEM-Beitrag ziemlich theorielastig, ging es doch um einen genetischen Schaltkreis für diagnostische Bakterien. Gewissermaßen sollten diese sich „merken“, was sie gesehen haben und später darüber berichten können. „Wir haben die Idee wirklich von der Logik und vom Modell her aufgezogen“, erinnert sich Mattia Gollub, Masterstudent der Computerwissenschaften, an den Beginn des Projekts. „Der Gedanke war, assoziatives Lernen in Bakterien zu implementieren.“
Begeistert vom Modell
Konditionierbare Bakterien – dazu fiel den Schweizern auch ein passender Projektname ein: Pavlov's Coli. Auch im jetzigen Gespräch merkt man noch, dass die Studenten eine große Begeisterung für genetische Schaltkreise und das Implementieren von Logik-Verknüpfungen auf DNA-Ebene einbrachten. Zuerst kam das theoretische Modell, dann das Tüfteln an den biologischen Komponenten – das schien die Herausforderung zu sein, die das Team reizte.
Einer der Team-Betreuer war übrigens Kobi Benenson, der an der ETH eine AG für Synthetische Biologie leitet und im April letzten Jahres auch im Rahmen eines Laborjournal-Artikels über Genetische Schaltkreise (4/2016: 32-36) zu Wort kam.
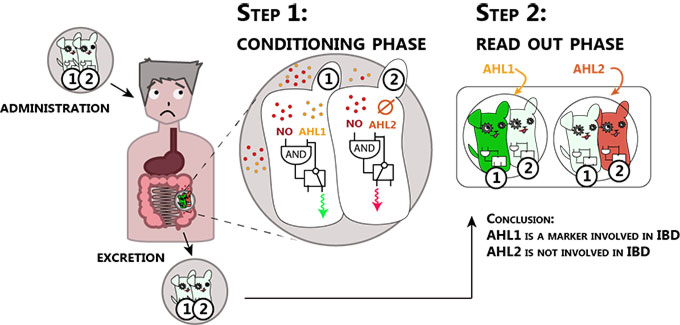
Nach dieser Skizze könnte die E. coli-Konditionierung auf Darm-reizende Mikroorganismen funktionieren.
Durch den Darm
Zunächst müssen die diagnostischen E. colis im Darm des Patienten jedoch erkennen, wann sie sich etwas einprägen sollen – und wann nicht. Nun ist die Sache mit den Darmerkrankungen nicht trivial: Der Darm eines gesunden Menschen beherbergt durchaus störungsfrei Bakterien, die der nächste Menschen mit anderem Mikrobiom nicht gut verträgt – so dass sie hier Entzündungen verursachen. Umgekehrt geht nicht jede Entzündung im Magen-Darm-Trakt auf ein gestörtes Mikrobiom zurück. In diesen Situationen sollen die Pawlow’schen E. colis natürlich keinen falschen Alarm schlagen.
Die Zürcher benötigten daher eine „UND-Verknüpfung“, wie der Informatiker sagt. Nur wenn zeitgleich eine Entzündung und ein charakteristischer Bakterienmarker vorliegen, dann sollten die diagnostischen Bakterien sich etwas einprägen. Als Entzündungsmarker diente letztlich Stickstoffmonoxid. Problematische Bakterien im Darm-Mikrobiom wollte das Team über spezifische AHL-Moleküle erkennen. „Das ist nicht ein bestimmtes Molekül, sondern eine ganze Klasse von Molekülen“, erklärt Lucas Schaus, der, wenn er nicht gerade in iGEM-Arbeit vertieft ist, seinen Biochemie-Master macht. „AHL steht allgemein für Acyl-Homoserin-Lactone“, fährt Schaus fort. „Und unterschiedliche Bakterienstämme produzieren unterschiedliche AHLs“.
Alternativ könnte solch ein Bakterienmarker auch ein spezifisches Lacton sein. „Normalerweise kommunizieren Bakterien über solche AHLs und Lactone“, beschreibt Schaus die natürliche Funktion der Moleküle. „Quorum Sensing“ nenne sich das, und Gemeinschaften von Mikroorganismen können so ihr jeweiliges Wachstum regulieren und abstimmen. In diesem Zusammenhang jedoch würden die AHLs den Pawlow’schen E. colis verraten, wenn etwas nicht stimmt.
Leserichtung ändern
Für ihr Testsystem verwendeten die Züricher das AHL eines Pflanzenpathogens. Für eine diagnostische Anwendung beim Menschen zwar uninteressant, aber, so Schaus, für die Proof of Concept-Versuche im Reagenzglas erstmal am einfachsten. „Dieses AHL ist in der Literatur beschrieben und bindet auch an unseren Transkriptionsrepressor EsaR“, begründet er die Wahl. Der Repressor unterdrückt die Expression einer Integrase. Das passende AHL entfernt diesen Repressor, so dass das Transkriptase-Gen abgelesen werden kann. Dazu braucht es allerdings noch die Anwesenheit des Entzündungsmarkers Stickstoffmonoxid, der ein an die DNA gebundenes Molekül namens NorR aktiviert. Erst dann exprimieren die E. colis tatsächlich die Integrase. Es müssen also beide Marker vorhanden sein – der Entzündungsmarker, wie auch derjenige für das „böse“ Bakterium!
„Integrasen können an sogenannte attP und attB-Sites auf der DNA binden“, erklärt Schaus, was weiter in den diagnostischen Bakterien passiert. „Dann dreht die Integrase die dazwischen liegende Sequenz um und ändern die Leserichtung.“ Weil die attP- und attB-Sites bei diesem Invertieren zerstört werden, lässt sich der Schalter nur ein einziges Mal umlegen. Dadurch „merken“ sich die E. colis gewissermaßen, was sie gesehen haben.

Das "Pavlov's Coli"-Team von der ETH Zürich (hinten rechts Mattia Gollub, vorne rechts Lucas Schaus)
Auf der invertierten Sequenz ist nun wiederum eine Bindestelle für den EsaR-Repressor angebracht – also den Repressor, der durch das AHL entfernt wird. Hier sitzt in der einen Leserichtung ein mNectarine-Gen, dessen Genprodukt orange Fluoreszenz bewirkt. In der anderen, nach Eingriff der Integrase relevanten Leserichtung liegt GFP. Das bedeutet: Nach dem Umdrehen der Sequenz leuchten die E. colis grün, wenn sie noch einmal demselben AHL begegnen. Folglich sind sie dann auf diesen Bakterienmarker konditioniert.
„Der Sinn dahinter ist, dass wir mehrere AHLs gleichzeitig messen können“, beschreibt Schaus die Langzeitvision. Dazu müsste man mehrere Repressoren entwickeln, die jeweils mit einem bestimmten AHL (oder einem anderen charakteristischen Pathogenmarker) interagieren und sich dann von der DNA lösen. So könnte man in ein und dasselbe Bakterium mehrere Konstrukte einbringen. Dieses „Multiplexing“ würde dann ermöglichen, dass man nach dem Weg durch den Darm die Pawlow’schen E. colis im Labor wachsen lässt und ihnen jeweils andere Marker präsentiert. Leuchtet E. coli orange, spielt der entsprechende Marker im Patienten keine Rolle. Leuchtet die Kultur aber grün, dann sind die E. colis diesem Marker schon mal im Zusammenhang mit einer Entzündung begegnet.
Theoretisch gibt auch andere Methoden, auf diese Art „Gelerntes“ auf der DNA abzuspeichern – zum Beispiel über CRISPR-basierte Methoden. Mehr dazu präsentiert das ETH-Team auf seinem Wiki.
Dass man „Pavlov’s Coli“ eines Tages tatsächlich die Diagnostik von Darmerkrankungen einsetzt, daran haben die Züricher derzeit noch Zweifel und bleiben zweckpessimistisch. Dazu müsse man zum einen mehr über das menschliche Mikrobiom und dessen Rolle bei Darmerkrankungen wissen, zum anderen müssten die Schaltkreise nicht nur im Modell, sondern auch im lebenden Kontext zuverlässiger funktionieren. Die Zeit habe gefehlt, alle Details wirklich auszutesten und zu optimieren.
Stolz ist Schaus aber auf die „AND-Gates“, die das Team entwickelt hat, also die Elemente, die logische UND-Verknüpfungen auf DNA-Ebene erlauben. „Das ist meiner Meinung nach das Innovativste an unserem Projekt – weil AND-Gates momentan ein limitierender Faktor sind. Wenn man nämlich mehrere AND-Gates in einer Zelle braucht, gehen einem irgendwann die Moleküle aus.“
Insgesamt konnten die Zürcher laut eigener Angaben 58 „Biobricks“ zum iGEM-Archiv beisteuern, also Plasmide mit Konstrukten, die andere Forscher und iGEM-Teams für eigene Arbeiten verwenden können. Darunter 18 AND-Gates. Daher wohl auch die Wettbewerb-Nominierung in der Kategorie „Best Part Collection“.
Mario Rembold
(Illustr. & Foto von http://2016.igem.org/Team:ETH_Zurich)
Mehr über weitere 2016er-iGEM-Teams aus dem deutschsprachigen Raum:
- Teams Aachen, Freiburg und München sowie Hintergrundinfos zu iGEM in unserer Dezember-Printausgabe (LJ 12/2016: 12-15)
- Team Hamburg im LJ online-Editorial vom 12.12.2016
- Team Düsseldorf im LJ online-Editorial vom 16.01.2017
- Team Bielefeld im LJ online-Editorial vom 30.01.2017
Letzte Änderungen: 27.02.2017










